SARS-CoV-2 . Agente etiologico, estructura y patogénesis.
Agente
etiológico:
Los
coronavirus pertenecen al Orden Nidovirales y Familia Coronaviridae. La Familia
Coronaviridae se reconoció por primera vez en 1968. Esta clasificación
taxonómica se asigna de acuerdo con la morfología, tipo de genoma y estrategia
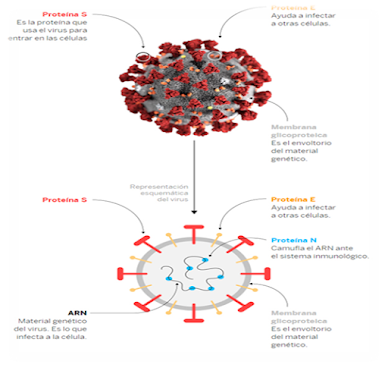
de replicación que presentan los virus de esta familia. El SARS-CoV-2 es un
beta coronavirus envuelto el cual contiene ARN de cadena sencilla. A estos
coronavirus al igual que el SARS-CoV-2 tienen una corona de puntas que se
observa alrededor del virus en imágenes de microscopía electrónica. Estas
puntas corresponden a las glicoproteínas espiga (S), distribuidas en toda la
superficie viral.
Estructura
Viral:
Como
hemos mencionado antes Los Coronavirus son virus envueltos de genoma ARN de una
sola cadena con un tamaño de entre 28 y 32 kb, con capside compleja y de envoltura
esférica que incluye unas espículas como una corona, de ahí el nombre de estos
tipos de virus. Las espículas están distribuidas simétricamente y son las que
permiten al virus abrirse camino por las membranas de las células y atacar a
éstas desde cualquier ángulo. El genoma contiene siete marcos de lectura
abiertos (ORF) que codifican para proteínas virales no estructurales. Presenta
una forma de corona cuando se observa al microscopio electrónico, la
glicoproteína S es el mediador de la unión y entrada de la célula huésped. El
ARN viral codifica 16 proteínas no estructuradas, que interfieren con la
respuesta inmune innata del hospedero. La parte restante del genoma del virus
codifica cuatro proteínas estructurales esenciales, incluida la glicoproteína
espiga (S), responsable de la unión y fusión del virus con las membranas
celulares; la proteína de membrana (M), responsable del transporte
transmembrana de nutrientes, liberación de la partícula viral y eventual
formación de su envoltura; las proteínas de nucleocápside (N) y las proteínas
de envoltura (E)
En
S1 se encuentra el dominio de unión al receptor (RBD, por sus siglas en inglés)
y es la parte más variable del genoma viral; mientras que S2 es una subunidad
altamente conservada, que media la fusión con la membrana celular y hace
posible su ingreso a la célula. Debido a que realiza una función indispensable,
la proteína S es el blanco principal de los anticuerpos neutralizantes.9,10 Al
comparar las características de SARS-CoV-2 y SARS-CoV se vio que comparten
únicamente el 40% de los aminoácidos del RBD y que la proteína S de SARS-CoV-2
tiene mayor afinidad por su receptor.10 La principal diferencia entre ambas
proteínas es una inserción de 12 nucleótidos entre S1 y S2, que forman un
motivo polibásico reconocido por furina. Se han encontrado mutaciones similares
en virus altamente patogénicos como el de la influenza, por lo que esto, junto
con la alta afinidad a su receptor, podrían ser la causa de la transmisión tan
eficiente del SARS-CoV-2.9
La
patogénesis de la infección por SARS-CoV-2 en humanos se da cuándo el virus accede
al tracto respiratorio a través de las membranas mucosas, especialmente la
nasal, orofaríngea y laríngea, una vez que ingresa a los pulmones por
continuidad. Luego puede ingresar a la sangre de manera periférica desde los
pulmones, lo que causa viremia19 y finalmente queda atacando todos los órganos
que expresan ECA220. Este es un receptor identificado como puerta de entrada
para SARS-CoV-2, que es una enzima que se adhiere a la membrana celular de
células ubicadas en el cerebro, corazón, arterias, endotelio respiratorio,
pulmones específicamente en sus células alveolares tipo II. Además el
SARS-CoV-2 se une a toda célula corporal que expresa ECA2 y TMPRSS en su
superficie, causando una respuesta inflamatoria sistémica. Esta se inicia con
una tormenta de citosinas, la cual consiste en una liberación de grandes
cantidades de citosinas pro inflamatorias (IFN-a, IFN-g, IL-1b, IL-6, IL-12,
IL-18, IL-33, TNF-a, TGFb, etc.) y quimosinas (CCL2, CCL3, CCL5, CXCL8, CXCL9,
CXCL10)23. La unión de SARS-CoV-2 a los
receptores ECA2 ubicados en la superficie de las AT2 reviste especial
importancia, ya que desencadena una cascada de inflamación en las vías
respiratorias inferiores, ocasionando un Síndrome de Dificultad Respiratoria
Aguda. La lesión característica de este síndrome, el daño alveolar difuso, ha
sido encontrada en la mayoría de pacientes fallecidos por neumonía grave por
COVID-19.
El
COVID-19 a lo largo de la pandemia se ha caracterizado por presentar
complicaciones como la miocarditis, la enfermedad tromboembolia, la coagulación
intravascular diseminada, la hipercitoquinemia y las complicaciones renales,
además de la enfermedad respiratoria.
El
SARS-CoV-2 no solo obtiene la entrada a las células pulmonares a través de
ECA2. Posteriormente, regula de forma negativa su expresión en la superficie de
estas células, a tal grado que la enzima se torna incapaz de ejercer efectos
protectores sobre los órganos corporales. La regulación negativa de la
expresión de ECA2 en las células pulmonares, conlleva a la acumulación sin
oposición de angiotensina II y a la activación local del sistema renina-angiotensina-aldosterona (SRAA).
Se ha postulado, pero no proba - do, que esta actividad no controlada de angiotensina
II puede ser en parte responsable de la lesión orgánica en COVID-1928,29, conllevando
a lesión pulmonar aguda, remodelación desfavorable del miocardio, vasoconstricción
periférica y permeabilidad vascular aumentada. Mientras que la activación local
del SRAA puede modular las lesiones pulmonares provocadas por la agresión
viral2







Muy bueno e interesante.
ResponderEliminar